着丝粒作为染色体上的枢纽区域,对于生物遗传信息的稳定性和精确传递起着决定性作用。然而,由于着丝粒DNA序列由大量高度相似的串联重复序列组成,其序列特征的精细解析一直是一个科学难题。
西安交大叶凯教授领导的信息与生物医学交叉团队,开发了针对基因组超复杂区域的计算方案,成功绘制了四种罂粟属物种的着丝粒序列图谱。这一些物种包括大红罂粟、虞美人、鸦片罂粟和渥美罂粟,它们各自拥有独特的核型特征。该团队开发混合组装技术,基于高精度长读长测序数据,克服了基因组超复杂区域的解析难题,为理解着丝粒在物种形成和演化中的作用提供了新视角。研究发现,着丝粒介导的染色体重排是形成物种复杂核型的关键机制,为生物重要性状的形成和稳定遗传提供了新的解释。
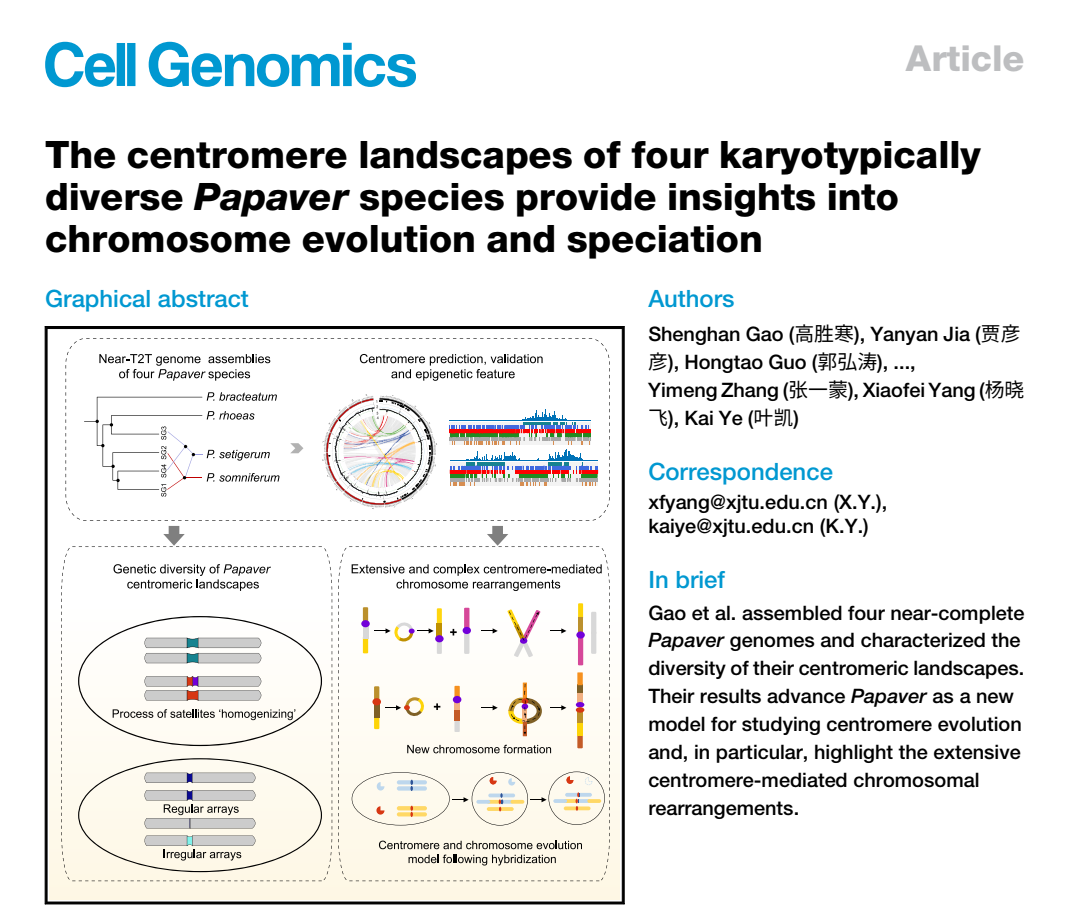
叶凯教授团队在罂粟属物种的研究领域取得了一系列重要成果。2018年,他们首次破译鸦片罂粟基因组,并在《科学》期刊上发表相关研究,发现吗啡合成通路超级基因簇。2021年,团队进一步探讨了该合成通路的演化,并在《自然-通讯》上发表了研究成果。最新的研究成果以"The centromere landscapes of four karyotypically diversePapaverspecies provide insights into chromosome evolution and speciation"为题,于7月30日在《细胞-基因组学》(Cell Genomics)在线发表。叶凯教授和计算机学院的杨晓飞教授担任论文的共同通讯作者,自动化学院的助理教授高胜寒和贾彦彦副研究员为论文的共同第一作者。
该研究得到了国家自然科学基金、科技部重点研发专项等项目的资助,展现了西安交大信息与生物医学交叉团队在多学科交叉创新、有组织科研方面的深厚实力。该团队致力于搭建多学科交叉科研平台,培养交叉学科人才,为我国AI赋能的生命健康和合成生物学领域等前沿生物领域的“卡脖子”问题提供原创解决方案。
信息与生物医学交叉研究中心团队主页:https://gr.xjtu.edu.cn/en/web/kaiye
论文链接:https://www.cell.com/cell-genomics/fulltext/S2666-979X(24)00230-1